Conteúdos
- Requisitos
- Objetivos da lição
- Introdução
- Um exemplo do dplyr em ação
- Operador Pipe
- O que é dplyr?
- Juntando tudo
- Conclusão
Requisitos
Nesta lição consideramos que já possui algum conhecimento da linguagem R. Se ainda não completou a lição Noções básicas de R com dados tabulares, recomendamos que o faça primeiro. Ter experiência com outras linguagens de programação também pode ser benéfico. Se está buscando por onde começar aprendendo outras linguagens, recomendamos os excelentes tutoriais de Python do Programming Historian.
Nota da tradução: o conteúdo da programação utilizado na lição original foi alterado para esta versão em português para que o contexto e os exemplos sejam próximos da realidade da comunidade que fala o idioma. Por conta disso, parte do texto da lição traduzida, bem como os exemplos e as interpretações dos dados são diferente da lição original. No entanto, o conteúdo e a estrutura da lição são fidedignos à lição original, como os tipos de dados e as análises desenvolvidas. Mudamos, por exemplo, a comparação entre Mississipi e Virgínia por Brasil e Argentina, mantendo os recursos e procedimentos realizados por Nabeel Siddiqui.
Objetivos da lição
Ao fim desta lição, você:
- Saberá como tornar seus dados bem ordenados (tidy) e entenderá por que isso é importante.
- Terá assimilado o uso do pacote dplyr (em inglês) e sua aplicação na manipulação e controle de dados.
- Estará familiarizado com o operador pipe
%>%na linguagem R e verá como ele pode auxiliar na criação de códigos mais legíveis. - Terá ganho experiência com análise exploratória de dados através de exemplos básicos de manipulação de dados.
Introdução
Os dados que encontra disponíveis nas diversas plataformas raramente estão no formato adequado para serem analisados, e precisará manipulá-los antes de explorar as perguntas de seu interesse. Isso pode tomar mais tempo que a própria análise dos dados! Neste tutorial, vamos aprender técnicas básicas para manipulação, gestão e controle de dados usando R. Especificamente, nos debruçaremos sobre a filosofia do “tidy data” (em inglês) conforme apresentada por Hadley Wickham.
De acordo com Wickham (em inglês), os dados estão tidy ou bem-organizados quando satisfazem três critérios chave:
- Cada unidade de observação está em uma linha
- Cada variável está em uma coluna
- Cada valor possui a sua própria célula.
Estar atento a estes critérios nos permite reconhecer quando os nossos dados estão adequados ou não. Também nos fornece um esquema padrão e um conjunto de soluções para lidar com alguns dos problemas mais comuns encontrados em datasets “mal-arranjados”, como por exemplo:
- Nomes de colunas como valores ao invés de nomes de variáveis
- Múltiplas variáveis contidas em uma única coluna
- Variáveis armazenadas tanto em linhas quanto em colunas
- Unidades de observação de diferentes categorias armazenadas na mesma tabela
- Uma única unidade de observação armazenada em múltiplas tabelas.
Talvez o mais importante seja que manter os dados nesse formato nos permite utilizar uma série de pacotes do “tidyverse,” (em inglês), concebidos para trabalhar especificamente com dados neste formato tidy. Dessa forma, assegurando-nos de que os dados de entrada e de saída estão bem organizados, precisaremos apenas de um pequeno conjunto de ferramentas para resolver um grande número de questões. Podemos combinar, manipular e dividir os datasets que criamos, conforme considerarmos mais adequado.
Neste tutorial focaremos no pacote dplyr (em inglês) presente no tidyverse, mas também é importante mencionar alguns outros que serão vistos na lição:
- magittr (em inglês) – Este pacote nos garante acesso ao operador pipe
%>%, que torna o nosso código mais legível. - ggplot2 (em inglês) – Este pacote utiliza a “Gramática de Gráficos” (em inglês) para fornecer uma forma fácil de visualizar nossos dados.
- tibble (em inglês) – Este pacote nos fornece uma releitura dos tradicionais data frames, mais fáceis de serem trabalhados e visualizados.
Instale o “tidyverse”, se ainda não o fez, e carregue-o antes de começarmos. Além disso, certifique-se de que possui instaladas a versão mais recente do R e a versão mais recente do RStudio compatíveis com o seu sistema operacional.
Copie o código a seguir para o seu RStudio. Para executá-lo, precisa selecionar as linhas e pressionar Ctrl+Enter (Command+Enter no Mac OS):
# Instala e carrega a biblioteca tidyverse
# Não se preocupe caso demore um pouco
install.packages("tidyverse")
library(tidyverse)
Um exemplo do dplyr em ação
Vejamos um exemplo de como o dplyr pode auxiliar historiadores. Vamos utilizar o pacote “dados” 1 e importar alguns indicadores socioeconômicos de países entre 1952 e 2007.
O pacote “remotes” permite a instalação de pacotes R a partir de repositórios remotos, incluindo o GitHub, como é o caso de “dados”.
# Instala e carrega as bibliotecas "remotes" e "dados"
install.packages("remotes")
library(remotes)
remotes::install_github("cienciadedatos/dados")
library(dados)
Em seguida, para termos acesso ao dataset “dados_gapminder”, que se encontra no pacote “dados”, basta executar o seguinte código:
# Cria o objeto dados_socioeconomicos_paises e atribui a ele os elementos de dados_gapminder
dados_socioeconomicos_paises <- dados_gapminder
Os dados do Gapminder (em inglês) contêm o progresso de países ao longo do tempo, observando as estatísticas de alguns índices. Após importar o dataset, notará que ele possui seis variáveis: país, continente, ano, expectativa de vida, população e PIB per capita. Os dados já estão em formato tidy, possibilitando uma infinidade de opções para exploração futura.
Neste exemplo, vamos visualizar o crescimento populacional de Brasil e Argentina ao longo dos anos. Para isso utilizaremos o pacote dplyr a fim de filtrar os dados que contenham apenas informações dos países de nosso interesse. Em seguida, utilizaremos o ggplot2 para visualizar tais dados. Este exercício é apenas uma breve demonstração do que é possível fazer com o dplyr, portanto, não se preocupe se não entender o código por enquanto.
# Filtra os países desejados (Brasil e Argentina)
dados_brasil_argentina <- dados_socioeconomicos_paises %>%
filter(pais %in% c("Brasil", "Argentina"))
# Visualiza a população dos dois países
ggplot(data = dados_brasil_argentina, aes(x = ano, y = populacao, color = pais)) +
geom_line() +
geom_point()
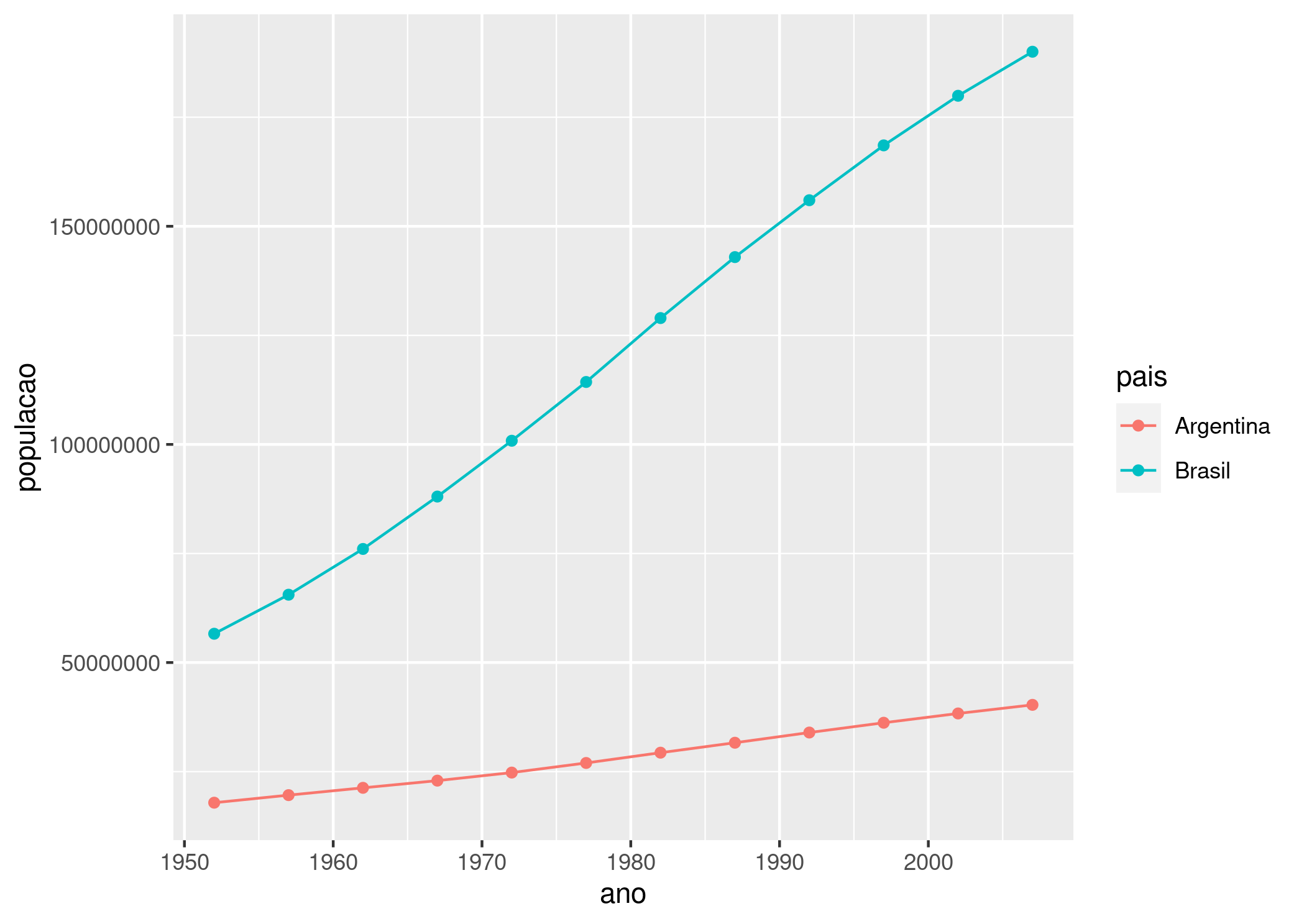
Gráfico da população de Brasil e Argentina, ao longo dos anos
Como podemos observar, a população absoluta do Brasil é consideravelmente maior em comparação com a população da Argentina. Embora isso pareça óbvio devido ao tamanho do território brasileiro, o código nos fornece uma base sobre a qual podemos formular uma infinidade de questões similares. Por exemplo, com uma pequena mudança no código podemos criar um gráfico similar com dois países diferentes, como Portugal e Bélgica.
# Filtra os países desejados (Portugal e Bélgica)
dados_portugal_belgica <- dados_socioeconomicos_paises %>%
filter(pais %in% c("Portugal", "Bélgica"))
# Visualiza a população dos dois países
ggplot(data = dados_portugal_belgica, aes(x = ano, y = populacao, color = pais)) +
geom_line() +
geom_point()
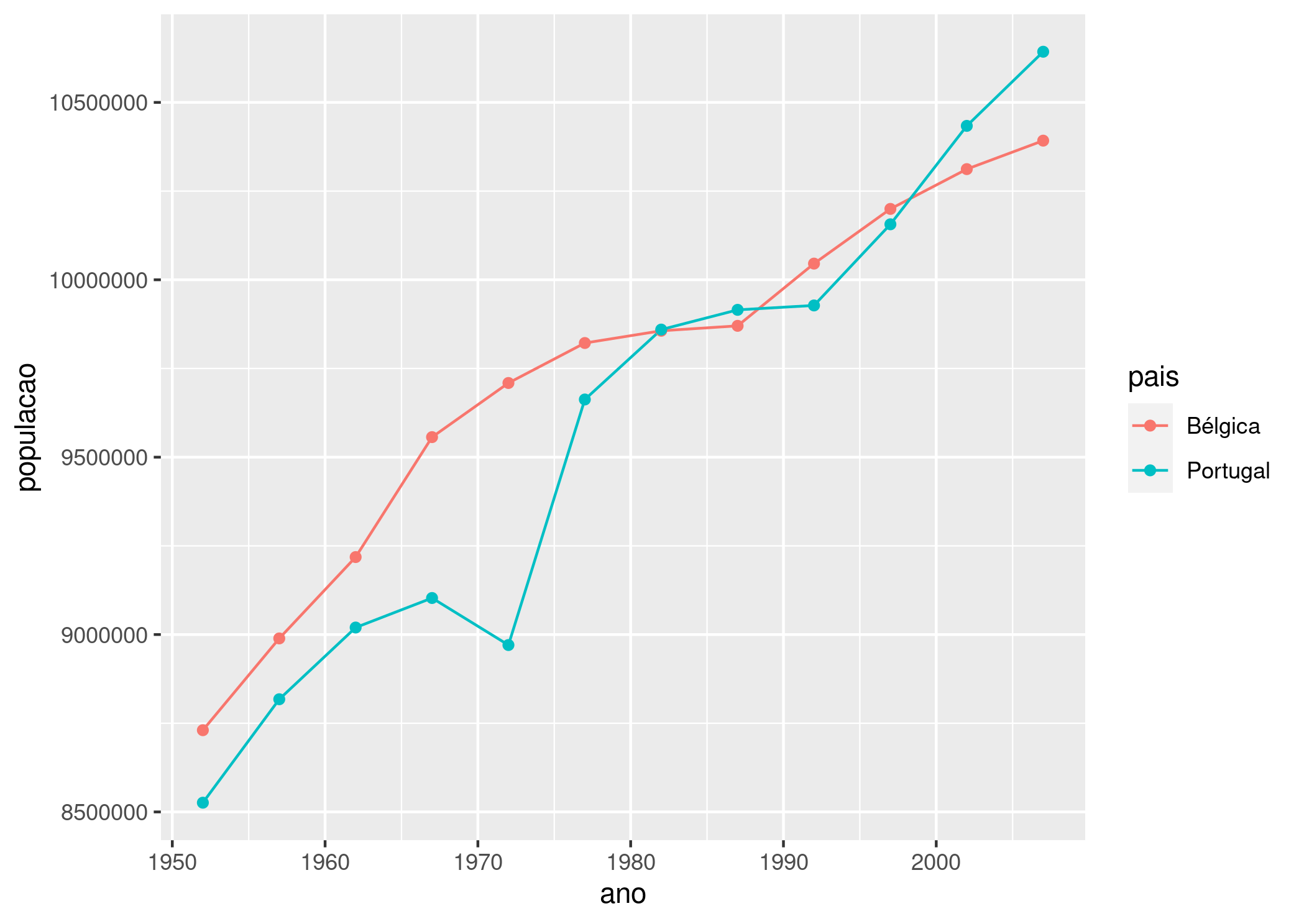
Gráfico da população de Portugal e Bégica, ao longo dos anos
Promover mudanças rápidas no código e revisar nossos dados é parte fundamental do processo de análise exploratória de dados (AED). Ao invés de tentar “provar” uma hipótese, a análise exploratória nos ajuda a entender melhor os dados e a levantar questões sobre eles. Para os historiadores, a AED fornece uma forma fácil de saber quando aprofundar mais em um tema e quando voltar atrás, e esta é uma área onde o R se sobressai.
Operador Pipe
Antes de olharmos para o dplyr, precisamos entender o que é o operador pipe %>% no R, uma vez que iremos utilizá-lo em muitos exemplos adiante. Como mencionado anteriormente, este operador é parte do pacote magrittr (em inglês), criado por Stefan Milton Bache e Hadley Wickham, e está incluída no tidyverse. O seu nome é uma referência ao pintor surrealista Rene Magritte, criador da obra “A Traição das Imagens”, que mostra um cachimbo com a frase “isto não é um cachimbo” (ceci n’est pas une pipe, em francês).
O operador pipe %>% permite passar o que está à sua esquerda como a primeira variável em uma função especificada à sua direita. Embora possa parecer estranho no início, uma vez que aprende a usar o pipe descobrirá que ele torna seu código mais legível, evitando instruções aninhadas. Não se preocupe se estiver um pouco confuso por agora. Tudo ficará mais claro à medida que observarmos os exemplos.
Vamos dizer que estamos interessados em obter a raiz quadrada de cada população e, então, somar todas as raízes antes de calcular a média. Obviamente, essa não é uma medição útil, mas demonstra a rapidez com que o código do R pode se tornar difícil de ler. Normalmente, usaríamos declarações aninhadas:
mean(sum(sqrt(dados_socioeconomicos_paises$populacao)))
## [1] 6328339
Veja que com tantos comandos aninhados fica difícil lembrar quantos parênteses são necessários no final da linha, tornando o código complicado de ler. Para atenuar esse problema, algumas pessoas criam vetores temporários entre cada chamada de função.
# Obtém a raiz quadrada da população de todos os países
vetor_raiz_populacao <- sqrt(dados_socioeconomicos_paises$populacao)
# Obtém a soma de todas as raízes da variável temporária
soma_vetor_raizes_populacao <- sum(vetor_raiz_populacao)
# Obtém a média da variável temporária
media_soma_vetor_raizes_populacao <- mean(soma_vetor_raizes_populacao)
# Exibe a média
media_soma_vetor_raizes_populacao
## [1] 6328339
Embora obtenha o mesmo resultado, este código é muito mais legível. No entanto, se esquecer de excluir os vetores temporários, seu espaço de trabalho pode se tornar confuso. O operador pipe faz esse trabalho por você. Aqui está o mesmo código usando o operador pipe:
dados_socioeconomicos_paises$populacao %>% sqrt %>% sum %>% mean
## [1] 6328339
Este código é mais fácil de ler que os anteriores e pode torná-lo ainda mais limpo escrevendo em linhas diferentes.
# Certifique-se de colocar o operador no final da linha
dados_socioeconomicos_paises$populacao %>%
sqrt %>%
sum %>%
mean
## [1] 6328339
Note que os vetores ou data frames criados pelo operador pipe são descartados quando se completa a operação. Se quiser salvar o resultado da operação, será preciso atribuí-lo a uma nova variável:
vetor_permanente_media_soma_populacao <- dados_socioeconomicos_paises$populacao %>%
sqrt %>%
sum %>%
mean
vetor_permanente_media_soma_populacao
## [1] 6328339
Agora que adquirimos uma compreensão do operador pipe, estamos prontos para começar a analisar e manipular alguns dados. Ao longo da lição vamos continuar trabalhando com o dataset dados_gapminder:
# Certifique-se de que o pacote "dados" está instalado e carregado aantes de proceder conforme abaixo
dados_gapminder
## # A tibble: 1,704 x 6
## pais continente ano expectativa_de_vida populacao pib_per_capita
## <fct> <fct> <int> <dbl> <int> <dbl>
## 1 Afeganistão Ásia 1952 28.8 8425333 779.
## 2 Afeganistão Ásia 1957 30.3 9240934 821.
## 3 Afeganistão Ásia 1962 32.0 10267083 853.
## 4 Afeganistão Ásia 1967 34.0 11537966 836.
## 5 Afeganistão Ásia 1972 36.1 13079460 740.
## 6 Afeganistão Ásia 1977 38.4 14880372 786.
## 7 Afeganistão Ásia 1982 39.9 12881816 978.
## 8 Afeganistão Ásia 1987 40.8 13867957 852.
## 9 Afeganistão Ásia 1992 41.7 16317921 649.
## 10 Afeganistão Ásia 1997 41.8 22227415 635.
## # … with 1,694 more rows
Como pode observar, este dataset contém o nome do país, seu continente e o ano de registro, além dos indicadores de expectativa de vida, total da população e PIB per capita, em determinados anos. Conforme mencionamos acima, antes de analisar os dados é importante verificar se estes estão bem ordenados no formato tidy. Relembrando os três critérios discutidos, podemos dizer que sim, o dataset encontra-se organizado e pronto para ser trabalhado com o pacote dplyr.
O que é dplyr?
Dplyr (em inglês) também é parte do tidyverse, fornecendo funções para manipulação e transformação dos dados. Porque estamos mantendo nossos dados bem organizados, precisaremos apenas de um pequeno conjunto de ferramentas para explorá-los. Em comparação com o pacote básico do R, usando o dplyr em nosso código, fica geralmente mais rápido e há a garantia de que os dados resultantes (output) estarão bem ordenados uma vez que os dados de entrada (input) também estarão. Talvez o mais importante seja que o dplyr torna o nosso código mais fácil de ser lido e utiliza “verbos” que são, na maioria das vezes, intuitivos. Cada função do dplyr corresponde a um desses verbos, sendo cinco principais: filtrar (filter), selecionar (select), ordenar (arrange), modificar (mutate) e sumarizar (summarise). Vamos observar individualmente como cada uma dessas funções funciona na prática.
Selecionar (select)
Se olharmos para o dataset dados_gapminder, vamos observar a presença de seis colunas, cada uma contendo diferentes informações. Podemos escolher, para a nossa análise, visualizar apenas algumas dessas colunas. A função select() do dplyr nos permite fazer isso. O primeiro argumento da função é o data frame que desejamos manipular e os seguintes são os nomes das colunas que queremos manter:
# Remove as colunas de dados_gapminder usando select()
# Note que não é necessário acrescentar o nome da coluna com o símbolo $ (dólar) ao final de dados_gapminder visto que o dplyr automaticamente assume que "," (vírgula) representa E (AND em inglês)
select(dados_gapminder, pais, ano, expectativa_de_vida)
## # A tibble: 1,704 x 3
## pais ano expectativa_de_vida
## <fct> <int> <dbl>
## 1 Afeganistão 1952 28.8
## 2 Afeganistão 1957 30.3
## 3 Afeganistão 1962 32.0
## 4 Afeganistão 1967 34.0
## 5 Afeganistão 1972 36.1
## 6 Afeganistão 1977 38.4
## 7 Afeganistão 1982 39.9
## 8 Afeganistão 1987 40.8
## 9 Afeganistão 1992 41.7
## 10 Afeganistão 1997 41.8
## # … with 1,694 more rows
Vejamos como escrever o mesmo código utilizando o operador pipe %>%:
dados_gapminder %>%
select(pais, ano, expectativa_de_vida)
## # A tibble: 1,704 x 3
## pais ano expectativa_de_vida
## <fct> <int> <dbl>
## 1 Afeganistão 1952 28.8
## 2 Afeganistão 1957 30.3
## 3 Afeganistão 1962 32.0
## 4 Afeganistão 1967 34.0
## 5 Afeganistão 1972 36.1
## 6 Afeganistão 1977 38.4
## 7 Afeganistão 1982 39.9
## 8 Afeganistão 1987 40.8
## 9 Afeganistão 1992 41.7
## 10 Afeganistão 1997 41.8
## # … with 1,694 more rows
Fazer referência a cada uma das colunas que desejamos manter apenas para nos livrar de uma é um tanto tedioso. Podemos usar o símbolo de menos (-) para demonstrar que queremos remover uma coluna.
dados_gapminder %>%
select(-continente)
## # A tibble: 1,704 x 5
## pais ano expectativa_de_vida populacao pib_per_capita
## <fct> <int> <dbl> <int> <dbl>
## 1 Afeganistão 1952 28.8 8425333 779.
## 2 Afeganistão 1957 30.3 9240934 821.
## 3 Afeganistão 1962 32.0 10267083 853.
## 4 Afeganistão 1967 34.0 11537966 836.
## 5 Afeganistão 1972 36.1 13079460 740.
## 6 Afeganistão 1977 38.4 14880372 786.
## 7 Afeganistão 1982 39.9 12881816 978.
## 8 Afeganistão 1987 40.8 13867957 852.
## 9 Afeganistão 1992 41.7 16317921 649.
## 10 Afeganistão 1997 41.8 22227415 635.
## # … with 1,694 more rows
Filtrar (filter)
A função filter() faz o mesmo que a função select, mas ao invés de escolher o nome da coluna, podemos usá-lo para filtrar linhas usando um teste de requisito. Por exemplo, se quisermos selecionar somente os registros dos países em 2007:
dados_gapminder %>%
filter(ano == 2007)
## # A tibble: 142 x 6
## pais continente ano expectativa_de_vida populacao pib_per_capita
## <fct> <fct> <int> <dbl> <int> <dbl>
## 1 Afeganistão Ásia 2007 43.8 31889923 975.
## 2 Albânia Europa 2007 76.4 3600523 5937.
## 3 Argélia África 2007 72.3 33333216 6223.
## 4 Angola África 2007 42.7 12420476 4797.
## 5 Argentina Américas 2007 75.3 40301927 12779.
## 6 Austrália Oceania 2007 81.2 20434176 34435.
## 7 Áustria Europa 2007 79.8 8199783 36126.
## 8 Bahrein Ásia 2007 75.6 708573 29796.
## 9 Bangladesh Ásia 2007 64.1 150448339 1391.
## 10 Bélgica Europa 2007 79.4 10392226 33693.
## # … with 132 more rows
Modificar (mutate)
A função mutate() permite adicionar uma coluna ao seu dataset. No momento, temos país e continente em duas colunas separadas. Podemos utilizar a função paste() para combinar as duas informações e especificar um separador. Vamos colocá-las em uma única coluna chamada “localizacao”.
dados_gapminder %>%
mutate(localizacao = paste(pais, continente, sep = ", "))
## # A tibble: 1,704 x 7
## pais continente ano expectativa_de_vida populacao pib_per_capita localizacao
## <fct> <fct> <int> <dbl> <int> <dbl> <chr>
## 1 Afeganistão Ásia 1952 28.8 8425333 779. Afeganistão, Ásia
## 2 Afeganistão Ásia 1957 30.3 9240934 821. Afeganistão, Ásia
## 3 Afeganistão Ásia 1962 32.0 10267083 853. Afeganistão, Ásia
## 4 Afeganistão Ásia 1967 34.0 11537966 836. Afeganistão, Ásia
## 5 Afeganistão Ásia 1972 36.1 13079460 740. Afeganistão, Ásia
## 6 Afeganistão Ásia 1977 38.4 14880372 786. Afeganistão, Ásia
## 7 Afeganistão Ásia 1982 39.9 12881816 978. Afeganistão, Ásia
## 8 Afeganistão Ásia 1987 40.8 13867957 852. Afeganistão, Ásia
## 9 Afeganistão Ásia 1992 41.7 16317921 649. Afeganistão, Ásia
## 10 Afeganistão Ásia 1997 41.8 22227415 635. Afeganistão, Ásia
## # … with 1,694 more rows
Novamente, é preciso lembrar que o dplyr não salva os dados, nem transforma o original. Em vez disso, ele cria um data frame temporário em cada etapa. Se deseja manter os dados, é necessário criar uma variável permanente.
dados_gapminder_localizacao <- dados_gapminder %>%
mutate(localizacao = paste(pais, continente, sep = ", "))
# Visualiza a nova tabela criada com a localização adicionada
dados_gapminder_localizacao
## # A tibble: 1,704 x 7
## pais continente ano expectativa_de_vida populacao pib_per_capita localizacao
## <fct> <fct> <int> <dbl> <int> <dbl> <chr>
## 1 Afeganistão Ásia 1952 28.8 8425333 779. Afeganistão, Ásia
## 2 Afeganistão Ásia 1957 30.3 9240934 821. Afeganistão, Ásia
## 3 Afeganistão Ásia 1962 32.0 10267083 853. Afeganistão, Ásia
## 4 Afeganistão Ásia 1967 34.0 11537966 836. Afeganistão, Ásia
## 5 Afeganistão Ásia 1972 36.1 13079460 740. Afeganistão, Ásia
## 6 Afeganistão Ásia 1977 38.4 14880372 786. Afeganistão, Ásia
## 7 Afeganistão Ásia 1982 39.9 12881816 978. Afeganistão, Ásia
## 8 Afeganistão Ásia 1987 40.8 13867957 852. Afeganistão, Ásia
## 9 Afeganistão Ásia 1992 41.7 16317921 649. Afeganistão, Ásia
## 10 Afeganistão Ásia 1997 41.8 22227415 635. Afeganistão, Ásia
## # … with 1,694 more rows
Ordenar (arrange)
A função arrange() nos permite ordenar as colunas de novas formas. Atualmente, o nosso conjunto de dados está organizado em ordem alfabética pelo nome do país. Vamos ordená-lo em ordem decrescente de acordo com o total da população.
dados_gapminder %>%
arrange(desc(populacao))
## # A tibble: 1,704 x 6
## pais continente ano expectativa_de_vida populacao pib_per_capita
## <fct> <fct> <int> <dbl> <int> <dbl>
## 1 China Ásia 2007 73.0 1318683096 4959.
## 2 China Ásia 2002 72.0 1280400000 3119.
## 3 China Ásia 1997 70.4 1230075000 2289.
## 4 China Ásia 1992 68.7 1164970000 1656.
## 5 Índia Ásia 2007 64.7 1110396331 2452.
## 6 China Ásia 1987 67.3 1084035000 1379.
## 7 Índia Ásia 2002 62.9 1034172547 1747.
## 8 China Ásia 1982 65.5 1000281000 962.
## 9 Índia Ásia 1997 61.8 959000000 1459.
## 10 China Ásia 1977 64.0 943455000 741.
## # … with 1,694 more rows
Sumarizar (summarise)
A última função do dplyr que veremos é a summarise(), usada geralmente para criar uma tabela contendo dados estatísticos resumidos que podemos plotar. Vamos utilizar a função summarise() para calcular a média da expectativa de vida nos países, considerando todo o conjunto dados_gapminder.
dados_gapminder %>%
summarise(mean(expectativa_de_vida))
## # A tibble: 1 x 1
## `mean(expectativa_de_vida)`
## <dbl>
## 1 59.5
Juntando tudo
Agora, após termos visto os cinco principais verbos do dplyr, podemos criar rapidamente uma visualização dos nossos dados. Vamos criar um gráfico de barras mostrando o número de países com expectativa de vida maior que 50 anos, em 2007.
expectativa_vida_2007 <- dados_gapminder %>%
filter(ano == 2007) %>%
mutate(expectativa_2007 = ifelse(expectativa_de_vida >= 50, "Maior ou igual a 50 anos", "Menor que 50 anos"))
ggplot(expectativa_vida_2007) +
geom_bar(aes(x = expectativa_2007, fill = expectativa_2007)) +
labs(x = "A expectativa de vida é maior que 50 anos?")
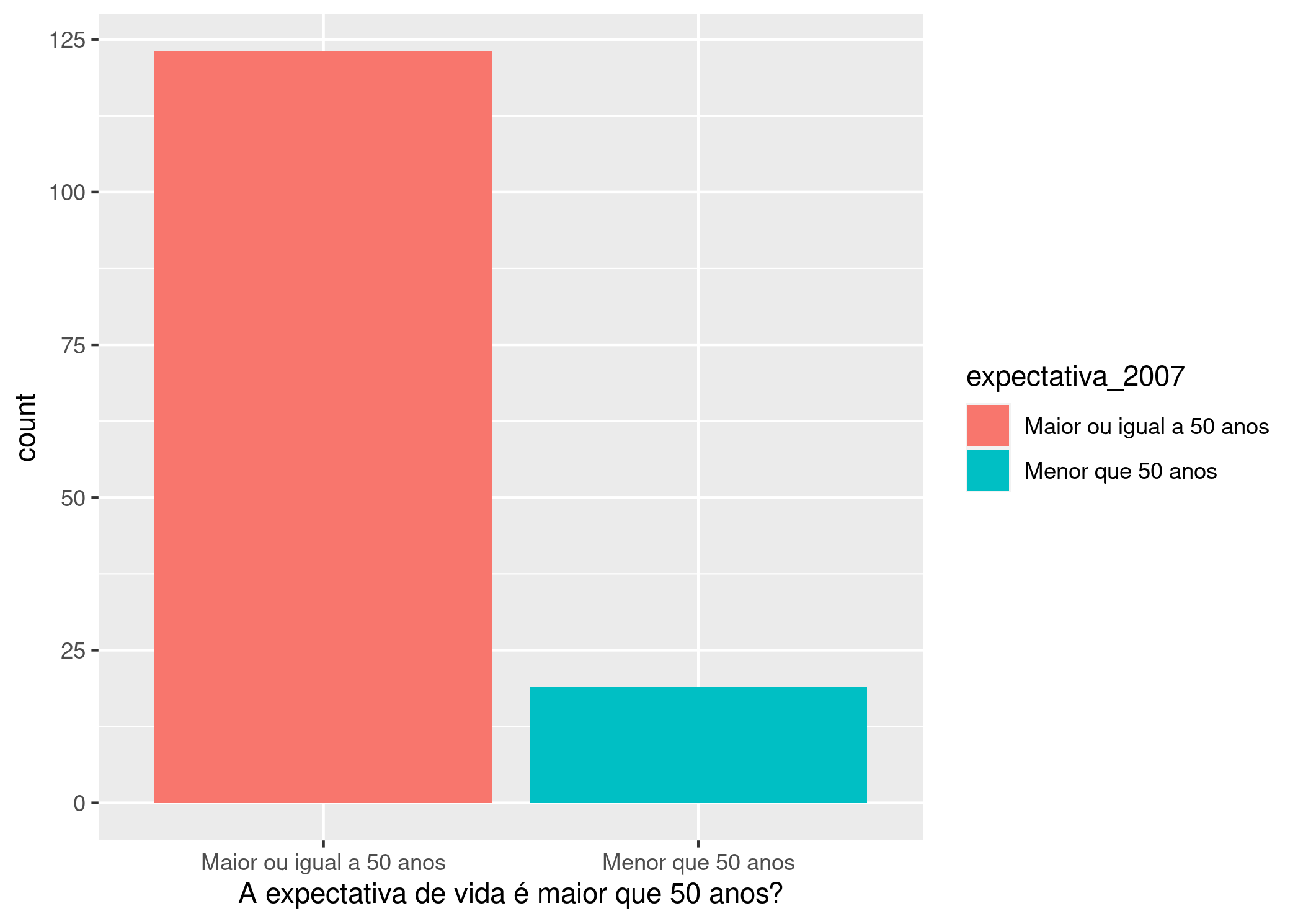
Expectativa de vida nos países em 2007
Novamente, fazendo uma pequena mudança no nosso código, podemos ver também o número de países com expectativa de vida maior que 50 anos, em 1952.
expectativa_vida_1952 <- dados_gapminder %>%
filter(ano == 1952) %>%
mutate(expectativa_1952 = ifelse(expectativa_de_vida >= 50, "Maior ou igual a 50 anos", "Menor que 50 anos"))
ggplot(expectativa_vida_1952) +
geom_bar(aes(x = expectativa_1952, fill = expectativa_1952)) +
labs(x = "A expectativa de vida é maior que 50 anos?")
(
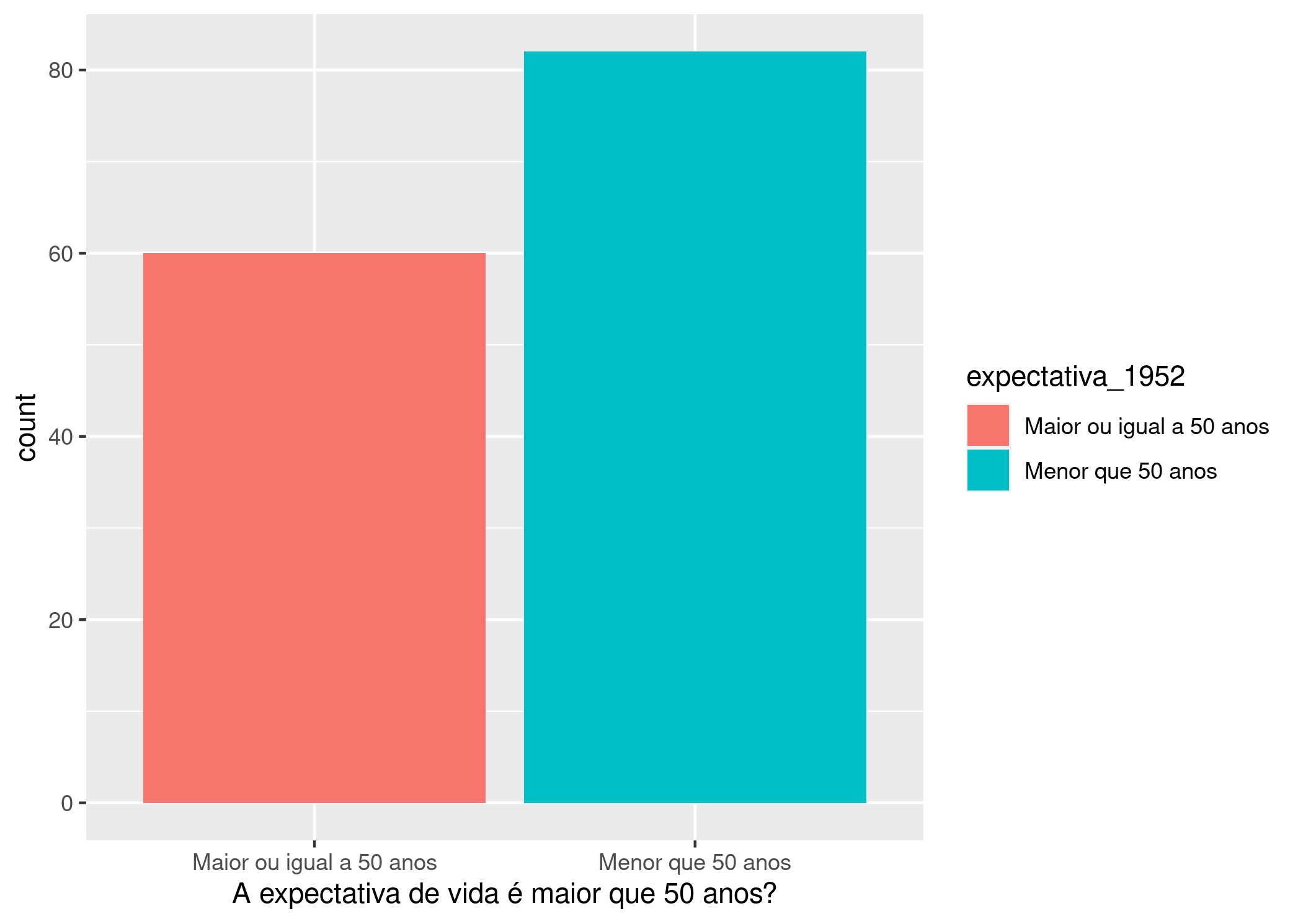
Expectativa de vida nos países em 1952
Conclusão
Este tutorial deve encaminhar seus conhecimentos para pensar sobre como organizar e manipular dados usando R. Posteriormente, provavelmente vai querer visualizar esses dados de alguma forma, usando gráficos, como fizemos em partes desta lição. Recomendamos que comece a estudar o ggplot2 (em inglês), pacote com uma coleção de ferramentas que funcionam bem em conjunto com o dplyr. Além disso, você deve buscar conhecer as outras funções do pacote dplyr que não vimos aqui, para aprimorar suas habilidades de manipulação de dados. Por enquanto, esta lição deve proporcionar um bom ponto de partida, cobrindo muitos dos principais problemas que poderá encontrar.
Notas
-
O pacote “dados” disponibiliza a tradução de conjuntos de dados originalmente em inglês encontrados em outros pacotes de R. Está disponível em https://github.com/cienciadedatos/dados ↩